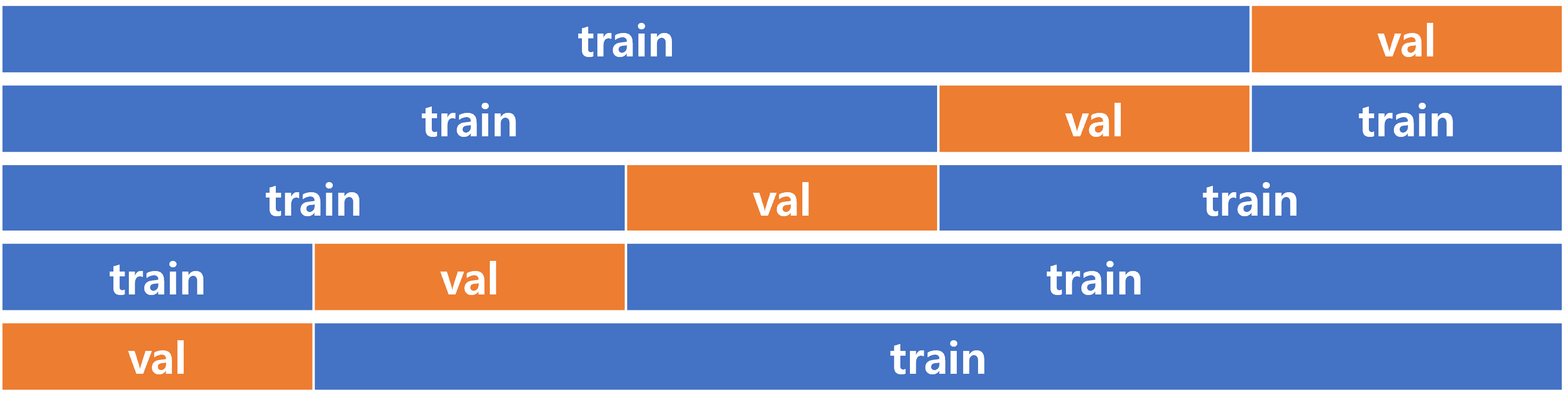
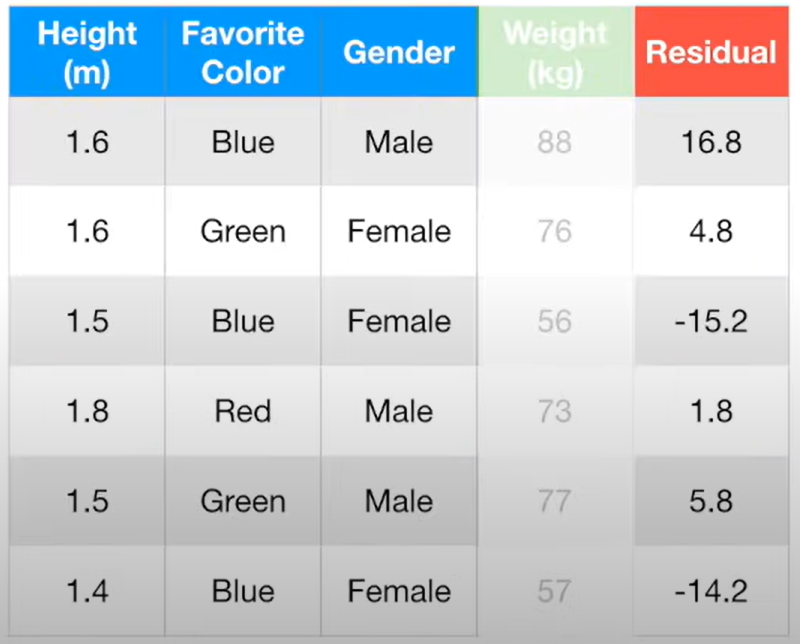
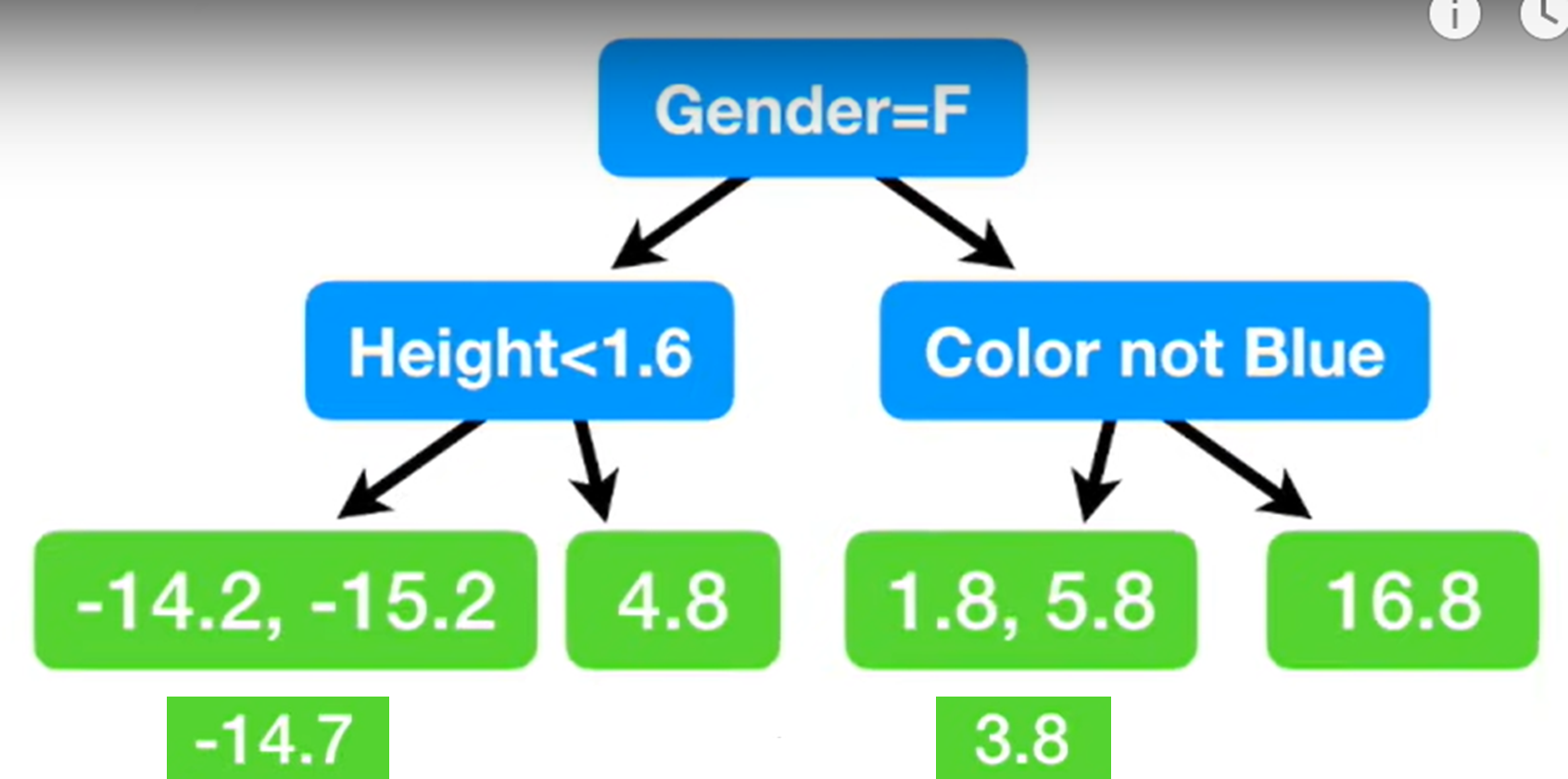
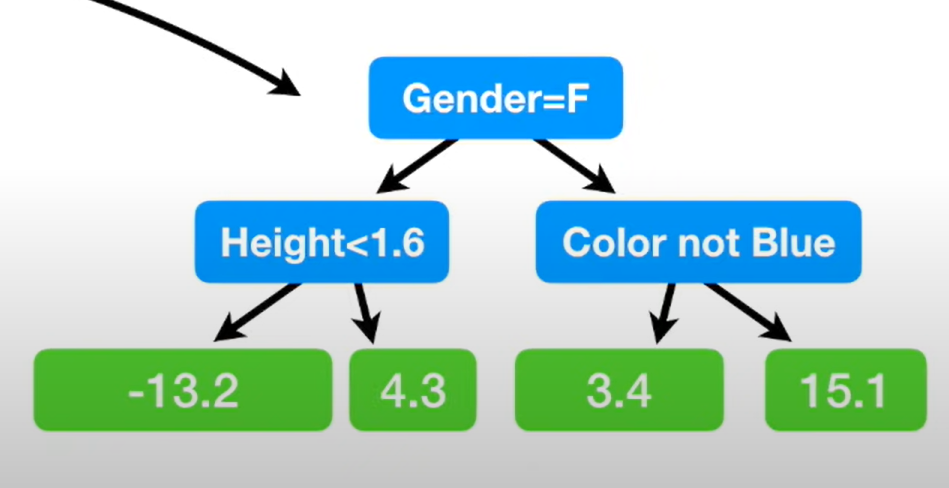
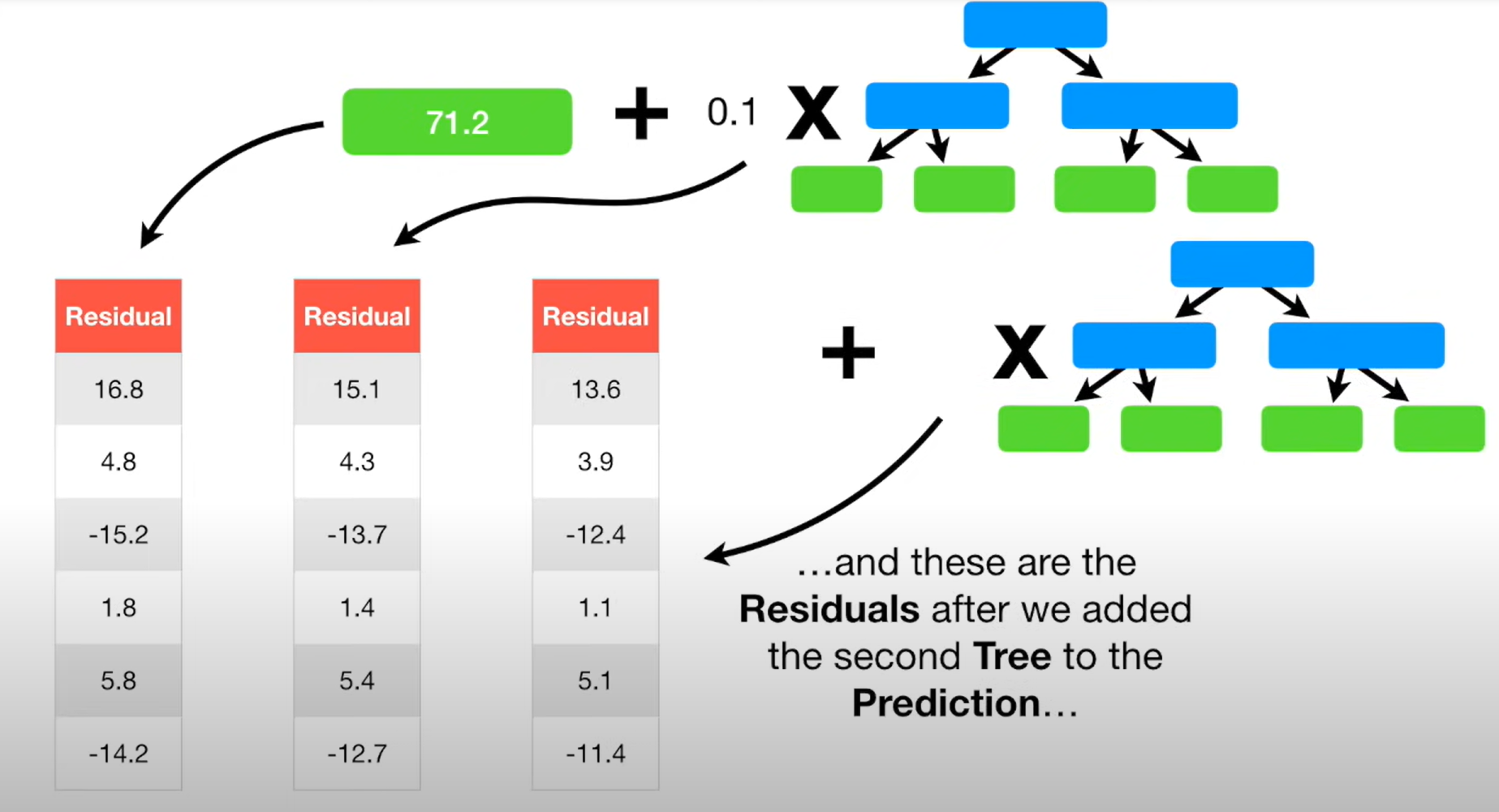
하이퍼파라미터 튜닝
1. 장점
- 다른 튜닝방법들이 존재하지만 상대적으로 빠르다고 할 수 있고
- 파라미터별 중요도를 직관적으로 확인할 수 있는 시각화까지 제공하여 용이하다
- 적용이 직관적이고 어렵지 않다는 장점 또한 뺴먹을수없다..
1. 모듈 임포트
import optuna
from optuna import Trial, visualization
from optuna.samplers import TPESampler
2. 목적값 정의
def lgbm_params_fold_start(lgb_params, X, y):
skf = StratifiedKFold(n_splits=5, shuffle=True, random_state=722)
skf.split(X, X['Gender'])
pred_list = []
mae_list = []
for fold,(train_index, val_index) in enumerate(skf.split(X, X['Gender'])):
print(f'***********{fold+1}th fold start***********')
x_train, x_val, y_train, y_val = X.loc[train_index], X.loc[val_index], y.loc[train_index], y.loc[val_index]
lgb = lightgbm.LGBMRegressor(**lgb_params, categorical_feature=[0])
lgb.fit(x_train, y_train,
eval_set=(x_val,y_val),
eval_metric=NMAE, verbose=False, early_stopping_rounds=100)
pred = lgb.predict(x_val)
result = NMAE(pred,y_val)[1]
mae_list.append(result)
return np.mean(mae_list)
먼저 최적화될 모델을 정의한다. 나는 5폴드의 lgbm을 정의했고 주목해야할 점은 lgb_params를 받아 학습을 실시하고 최적화할 해를 리턴값으로 리턴하는다는점이다. 폴드를 적용하지 않는다면 objective에 바로 모델을 넣어도 무방하다.
def objectiveLGB(trial: Trial, X, y):
param = {
'boosting_type' : 'gbdt',
"n_estimators" : 10000,
# 'fold_size':trial.suggest_int('fold_size', 4, 16),
'max_depth':trial.suggest_int('max_depth', 4, 16),
'random_state': 722,
'reg_alpha': trial.suggest_loguniform('reg_alpha', 1e-8, 10.0),
'reg_lambda': trial.suggest_loguniform('reg_lambda', 1e-8, 10.0),
'num_leaves': trial.suggest_int('num_leaves', 8, 32),
'colsample_bytree': trial.suggest_uniform('colsample_bytree', 0.5, 1.0),
'subsample': trial.suggest_uniform('subsample', 0.8, 1.0),
'subsample_freq': trial.suggest_int('subsample_freq', 1, 8),
'min_child_samples': trial.suggest_int('min_child_samples', 16, 64),
'learning_rate': 0.025
}
score = lgbm_params_fold_start(param, X, y)
return score이후 파라미터의 범위를 지정하는 objective함수를 정의한다.
딕셔너리 형태로 정의하며 각 파라미터의 특성에 맞게 아래의 것으 선택하면되겠다.
- suggest_int : 범위 내의 정수형 값
ex) 'max_depth':trial.suggest_int('max_depth', 4, 16)
- suggest_int : 범위 내의 실수형 값
ex) 'max_depth':trial.suggest_float('max_depth', 4, 16)
* 위 두 방법은 log=True 메소드를 통해 로그값도 가능
- suggest_categorical : list 내의 데이터 선택
ex) 'colsample_bytree': trial.suggest_categorical('colsample_bytree', [0.5,0.6,0.7])
- suggest_uniform : 범위내의 균등분포를 값으로 선택
ex) 'colsample_bytree': trial.suggest_uniform('colsample_bytree', 0.5, 1.0)
- suggest_discrete_uniform : 범위내의 이산균등분포를 값으로 선택
ex) 'colsample_bytree': trial.suggest_uniform('colsample_bytree', 0.5, 1.0, 0.1)
- suggest_loguniform : 범위 내의 로그함수 선상의 값을 선택
ex) 'reg_lambda': trial.suggest_loguniform('reg_lambda', 1e-8, 10.0)
def objective(trial):
x, y = ...
classifier_name = trial.suggest_categorical("classifier", ["SVC", "RandomForest"])
if classifier_name == "SVC":
svc_c = trial.suggest_float("svc_c", 1e-10, 1e10, log=True)
classifier_obj = sklearn.svm.SVC(...")
else:
rf_max_depth = trial.suggest_int("rf_max_depth", 2, 32, log=True)
classifier_obj = sklearn.ensemble.RandomForestClassifier(...)
accuracy = ...
return accuracy위 와같은 방법으로 모델 선택도 가능
3. 실행
study = optuna.create_study(direction='minimize',sampler=TPESampler(seed=42))
study.optimize(lambda trial : objectiveLGB(trial, X, y), n_trials=100)
print('Best trial: score {},\nparams {}'.format(study.best_trial.value,study.best_trial.params))study 라는 객체를 생성하여 실행한다.
- direction 은 목적한 objective 함수를 최소화할 것인지 최소화할것인지 입력한다 minimize, maximize
- sampler는 어떻게 값을 찾을 것인지 결정한다.
https://optuna.readthedocs.io/en/stable/reference/samplers.html
객체의 optimize 메소드를 활용해 튜닝을 시작한다.
- n_trials 는 시도횟수를 의미한다.
완료되면 study best_trial.value 메소드를 이용하여 목적함수의 값
best_trial.params 메소드를 이용해 파라미터들의 값을 받을 수 있다.
4. 시각화
optuna.visualization.plot_param_importances(study)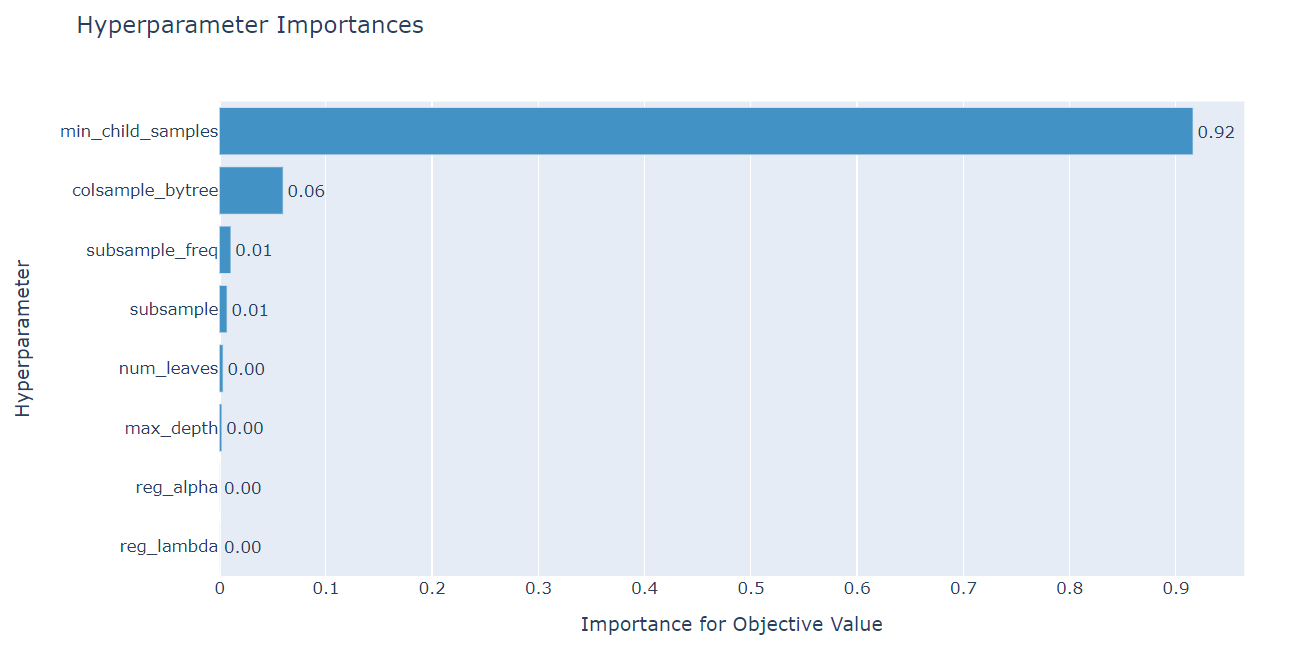
각 파라미터의 중요도를 나타내는 그래프
optuna.visualization.plot_optimization_history(study)

최적화 과정을 보여주는 그래프
'ML' 카테고리의 다른 글
| [ML] 교차검증, cross-validation (0) | 2022.04.11 |
|---|---|
| [ML] 인공신경망 학습 (0) | 2022.02.17 |
| [ ML ] 인공신경망 기본 (0) | 2022.02.16 |
| [ML] cycling encoding (0) | 2022.02.15 |
| [ML] XGBoost - Classification (0) | 2022.01.18 |