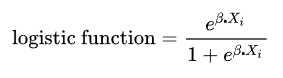from pmdarima.arima import auto_arima
auto_arima_result = auto_arima(airDF, start_p = 1, start_q = 1, max_p = 3, max_q = 3,
seasonal = True, d = 1, D=1, m=12, start_P = 1, start_Q = 1, max_P = 3, max_Q = 3, trace=True,
error_action = 'ignore', suppress_warnings = True, stepwise=False)1. 시계열
통계학에서 시계열 데이터 분석은 a변수로 b변수를 예측하는 것이 아닌 b변수의 과거데이터들을 이용하여 미래 시점을 예측하게 된다. t-1,...,t-n 시점의 데이터들을 이용해서 t시점의 데이터들을 예측하게 된다.
2. 분해법
데이터
airDF = pd.read_csv('international-airline-passengers.txt',
parse_dates=['time'], index_col='time')
airDF.head(3)| time | passengers |
| 1949-01-01 | 112 |
| 1949-02-01 | 118 |
| 1949-03-01 | 132 |
월별 주기로 이루어져있는 데이터
from statsmodels.tsa.seasonal import seasonal_decompose
result = seasonal_decompose(airDF, freq=12, model = 'addictibve')
# result = seasonal_decompose(airDF-1, freq=12, model = 'multiplicativbe')
result.plot()
plt.show()
# result.observed 관찰값
# result.trend 추세성분
# result.seasonal 계절성분
# result.resid 잔차
분해법은 시계열의 성분이 어떠한 패턴을 가지고 이루어져있을 것이라는 가정하에
Zt = Tt (추세성분 : 장기적인 변동) +
St (계절성분 : 주별, 월별, 분기별 처럼 주기가짧은 규칙적으로 반복되는 변동)+
Ct (순환성분 : 계절성분과 유사하다나 구기가 김, 많은 경우 분해법을 적용할 때 포함하지 않ㅇ므) +
It (불규칙성분 )
위처럼 분해하여 설명하는 모형이다. 각 성분을 잇는것이 +이냐 * 이냐에 따라
가법모형과 승법모형으로 나뉘게 된다 .
3. ARIMA
acf, pacf
from statsmodels.graphics.tsaplots import plot_acf, plot_pacf
def plot_ts_corr(ts):
fig, axes= plt.subplots(1,2, figsize = (10, 4))
plot_acf(ts, ax = axes[0], lags = 30)
plot_pacf(ts, ax = axes[1], lags = 30)
plt.show()
plot_ts_corr(airDF)
자기상관계수 : 자신의 t시점차이의 데이터와의 상관계수 x축은 시점 y축은 상관계수값
부분자기상관계수 : 자신의 t시점차이의 데이터와의 상관계수 단, t시점과 시차가 n인 상관계수값은 시차가 1인 데이터 시차가 2인데이터 ... 시차가 n-1 인데이터들의 영향을 제거하여 계산
x축은 시점 y축은 상관계수값
위 그래프를 보고 arima모델의 pdq를 예측함
정상성
arima 모델을 적용하려면 정상시계열이라는 조건이 필요하다.
정상시계열이란
E(Xt)가 t의 값과 상관없이 일정
Var(Xt)가 존재하고 t의 값에 상관없이 일정
Cov(Xt, Xt+h)가 t와는 무관하고 h(시차)의 값에만 의존
위 조건을 만족하는 시계열 데이터이다.

예시 데이터는 진폭이 일정하지않고 (분산이 일정하지 않고) 평균값이 시차에 의존하는것으로 보이기 때문에 비정상시계열일 것이다. 추가적으로 정상성을 만족하는지 확인하는 검정과정을 ㅅ행한다
from statsmodels.tsa.stattools import adfuller
def adfuller_test(ts):
test = adfuller(ts, maxlag=12)[:2]
print('*** adfuller-test *** \n alternative hypothesis: stationary \n statistic = %6.3f p-value = %6.4f\n' % test)
adfuller_test(airDF)*** adfuller-test ***
alternative hypothesis: stationary
statistic = 1.565 p-value = 0.9977pvalue가 아주 높으므로 정상성을 띈다는 대립가설을 기각한다. 즉 위의 데이터는 비정상 시계열이다.
정상성을 만족하게 하는 방법은 여러가지가 있지만 보통 차분을 사용한다.
fig, axes= plt.subplots(2,1, figsize = (10, 6))
axes[0].plot(airDF, color = 'red')
axes[0].legend(['before diff'])
axes[1].plot(airDF.diff(1))
axes[1].legend(['after diff'])
plt.show()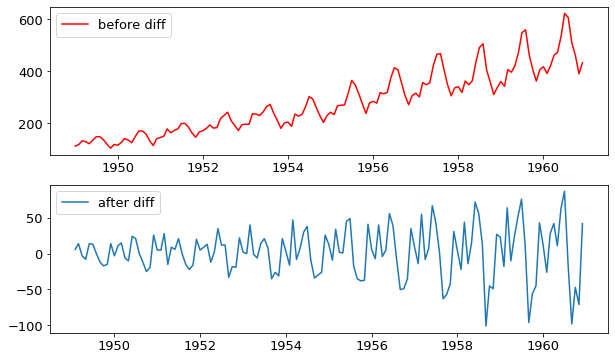
차분 이후에도 분산이 점점 시간이 지남에 따라 커지는 것으로 보아 아직 정상시계열이 아닌것으로 보이지만 위와 같은 방법으로 차분을 실시할 수 있다.
| Model | AR(p) | MA(q) | ARMA(p,q) |
| ACF | 지수적 감소 | q시점 이후로 절단 | 지수적 감소 |
| PACF | P시점 이후로 절단 | 지수적 감소 | 지수적 감소 |
시계열 데이터가 정상시계열이라는 가정하에 (차분을 거치며 테스트로 확인 차분횟수 = d)
위의 표의 기준으로 차수를 결정이후
from statsmodels.tsa.arima.model import ARIMA
arima_res = ARIMA(airDF, order=(1, 1, 0)).fit()
arima_res.summary()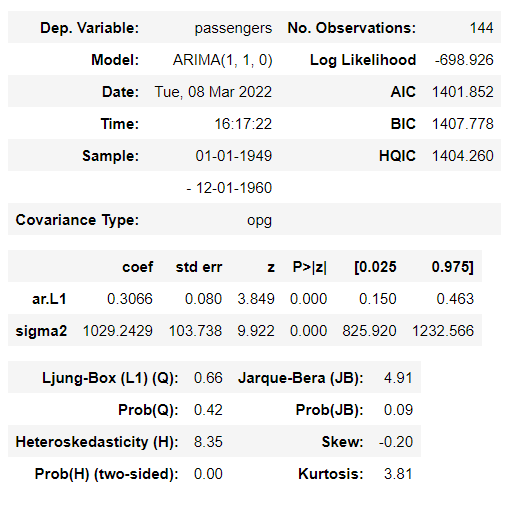
위와 같은 결과를 얻을 수 있다. order 이 1,1,0 이라는 의미는 한번 차분이후 p=1 q=0이라는 의미이고
Yt = 0.3Yt-1 + e
이런 모형이고 sigma2는 e잔차의 분산값이다.
def forecast_plot(data, fit, num):
try:
pred = fit.get_forecast(num)
pred_val = pred.predicted_mean
pred_lb = pred.conf_int().iloc[:,0]
pred_ub = pred.conf_int().iloc[:,1]
pred_index = pred_ub.index
except:
arima_res = ARIMA(airDF, order=(1, 0, 0)).fit()
pred22 = arima_res.get_forecast(num)
pred_index = pred22.predicted_mean.index
pred = fit.predict(num, return_conf_int =True)
pred_val = pred[0]
pred_lb = pred[1][:,0]
pred_ub = pred[1][:,1]
fig, axes= plt.subplots(figsize = (10, 4))
axes.plot(data)
axes.vlines(pred_index[0],0,700, linestyle='--', color = 'red')
axes.plot(pred_index,pred_val, label ='predict')
axes.fill_between(pred_index, pred_lb, pred_ub, color ='k', alpha=0.1, label='0.95 interval')
plt.title('prediction')
plt.show()
forecast_plot(airDF,arima_res,12)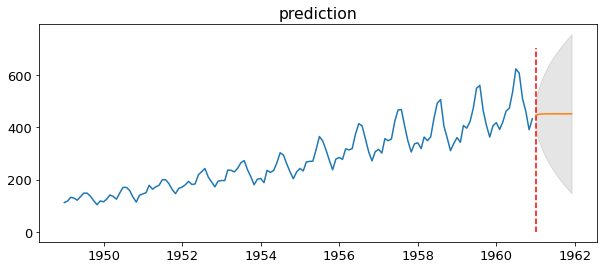
주황값이 예측값이다.
위의 데이터는 계절성이 있지만 위 arima모델로는 계절성을 설명하지 못하기때문에 옳지 못하다. 그렇기에 후에 Sariama모델을 설명핟겓.
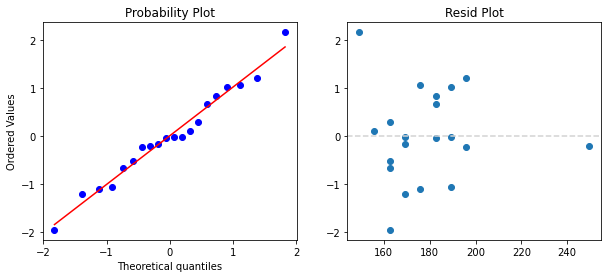
arima_res.plot_diagnostics()
plt.show()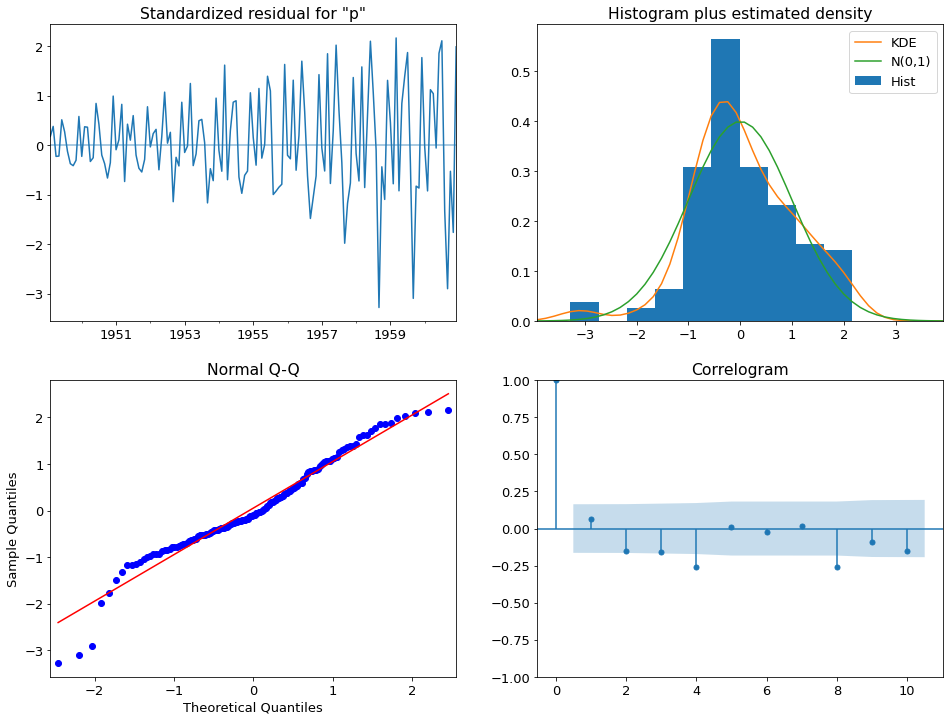
원하는 결과가 나왔다면 백색잡음과정의 ㄱ가정을 몇가지 확인하게 된다.
E(Xt) = 0
Var(Xt) = sigma^2 (all t)
Cov(Xt, Xs) = 0 (t!=s)
정규분포의 갖어과 비슷하지만 iid의 가정이 빠진경우이다.
위가정이 만족하는지 보여주는 그래프들을 보며 가정이 맞는지 확인한다.
하지만 좌측상단의 그래프를 보면 분산이 t가 커짐에 따라 커지는 것을 확인할 수 있기때문의 위의 적합은 옳지 않다고 할 수 있다.
from statsmodels.tsa.statespace.sarimax import SARIMAX
sarima_res = SARIMAX(airDF, order=(1, 1, 0), seasonal_order = (1,1,2,12)).fit()
sarima_res.summary()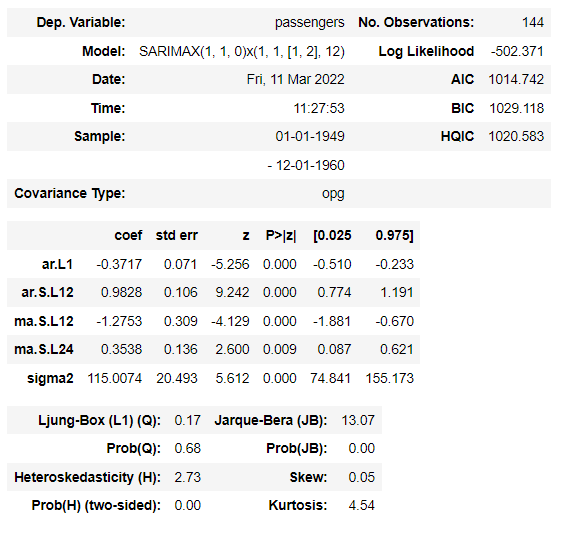
계절성의 주기가 12라는 뜻이다. seasonal_order은 계절차수이다.
forecast_plot(airDF,sarima_res,12)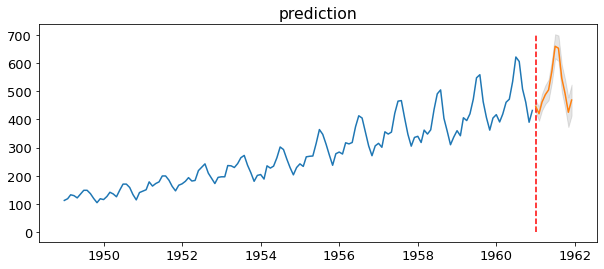
이상적이어 보인다.
sarima_res.plot_diagnostics()
plt.show()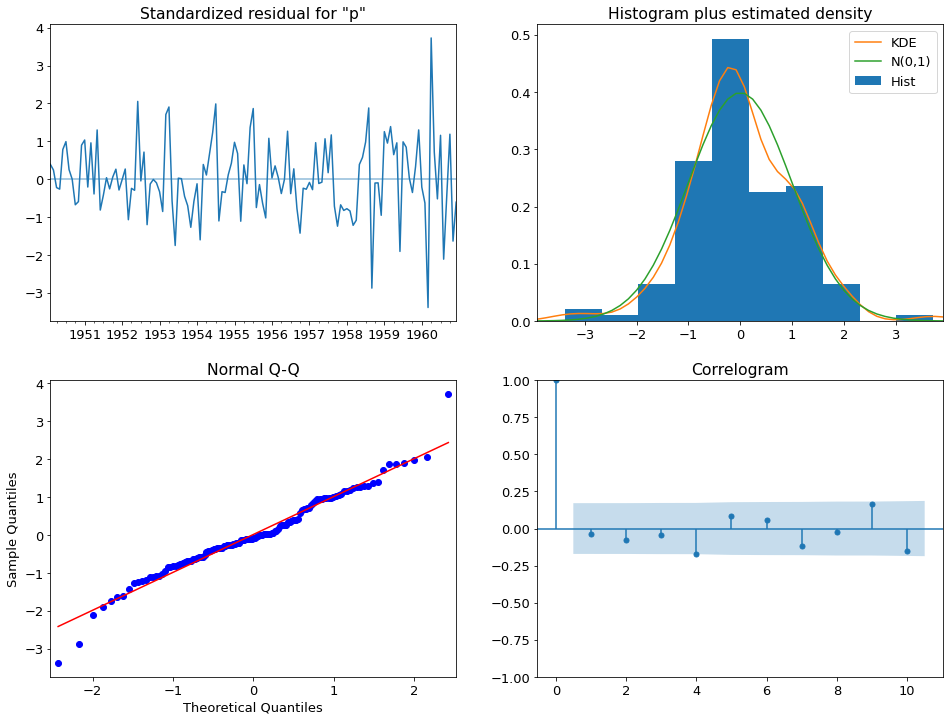
가정이 아주 잘 만족하는 것으로 보인다.
from pmdarima.arima import auto_arima
auto_arima_result = auto_arima(airDF, start_p = 1, start_q = 1, max_p = 3, max_q = 3,
seasonal = True, d = 1, D=1, m=12, start_P = 1, start_Q = 1, max_P = 3, max_Q = 3, trace=True,
error_action = 'ignore', suppress_warnings = True, stepwise=False)원래는 차분을 반복하며 어떤 차수가 맞는지 반복하고 acf,pacf에 맞추어 p와 q를 맞추는 과정이 필요하지만
위의 모듈을 사용하면 자동적으로 aic가 낮은 녀석을 선택해준다.
위의 가정을 확인하는 함수나 예측시각화 그래프, summary 또한 그대로 적용가능하다
'통계' 카테고리의 다른 글
| [통계] 로지스틱 회귀분석 (0) | 2022.03.10 |
|---|---|
| [통계] 단순선형회귀분석, 모형진단, 영향점분석 (0) | 2022.03.04 |
| [통계] 상관분석 (0) | 2022.03.03 |
| [통계] ANOVA 분산분석 (0) | 2022.02.23 |
| [통계] t-test (0) | 2022.02.23 |